细胞游离DNA(cfDNA)检测整体研究方案
CfDNA testing protocol
细胞游离DNA(cell-free DNA,cfDNA)是指在生物体的体液中(如血浆、尿液、脑脊液等)自由存在的、非细胞内的DNA片段。这些DNA片段通常来源于细胞凋亡(程序性死亡)或坏死(细胞损伤或死亡后释放),可以被释放到循环系统中,并在体液中被检测到。cfDNA研究和应用是精准医疗和液体活检领域的重要方向之一。
cfDNA特点:来源多样、片段短小、半衰期短、非侵入性检测
cfDNA应用领域:肿瘤学、产前诊断、移植医学、移植性疾病等
cfDNA检测研究方案
1. 研究目标
- 确定cfDNA在特定疾病(如肿瘤学、产前诊断等)早期筛查和诊断中的有效性和准确性。
- 识别和验证疾病特异性的cfDNA甲基化和羟甲基化标记。
- 评估cfDNA组蛋白修饰在疾病早期诊断中的作用。
2. 研究设计
- 队列选择:选择特定疾病高风险人群和健康对照组,确保样本的代表性和多样性。
- 样本收集:收集血浆、尿液、脑脊液等样本,并确保样本的质量和存储条件符合要求。
3. 检测技术
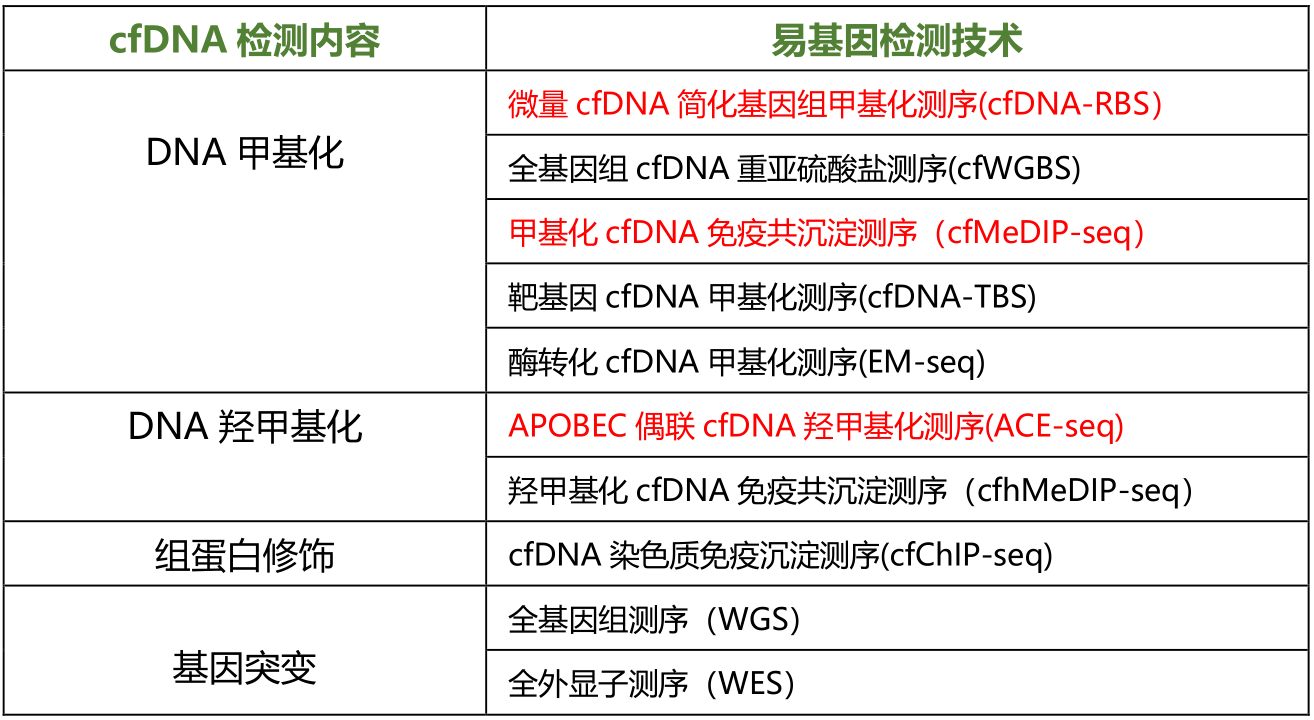
- cfDNA甲基化检测:使用cfDNA-RBS、cfWGBS、cfMeDIP-seq等技术进行甲基化模式分析。
- cfDNA羟甲基化检测:采用ACE-seq和cfhMeDIP-seq技术进行羟甲基化水平分析。
- cfDNA组蛋白修饰检测:利用cfChIP-seq技术研究组蛋白修饰水平。
- cfDNA基因突变检测:通过WGS和WES技术检测cfDNA基因组变异。
4. 数据分析
- 生物信息学分析:对测序数据进行质量控制、比对、变异检测和甲基化水平分析。
- 统计分析:使用机器学习模型识别疾病特异性标记,并评估其诊断效能。
5. 验证和临床应用
- 验证研究:在独立队列中验证发现的生物标志物。
- 临床应用:与临床数据结合,评估cfDNA检测在实际临床诊断中的应用价值。
cfDNA检测样本选择和送样要求
样本选择
· 血浆样本:血浆能避免所含血细胞裂解引起的DNA污染,是cfDNA检测的优先选择。
· 脑脊液样本:通常作为中枢神经系统疾病研究的样本选择。
· 尿液样本:常用于泌尿系统癌症如膀胱癌、肾癌等肿瘤标志物研究及移植排异监测等cfDNA研究。
血浆样本要求
· 样本采集:静脉抽血5-10ml,收集到EDTA抗凝血管中,上下颠倒抗凝采血管,轻轻混匀,全血2-8℃保存不超过4h;如用Streck采血管收集全血,则5-8℃保存不超过72h。
· 分离血浆:采血后建议马上分离血浆,提取cfDNA,-20℃保存;如不能马上提取,常规采血管采血后必须4h内(最好30min-1h内)完成血浆分离,-80℃保存。
· 样本运输:大体积干冰运输。
cfDNA研究案例
(1)ctDNA全基因组亚硫酸盐测序分析,用于癌症检测和分子分类
此研究纳入三组乳腺癌(BC)患者(BC组n=123;健康对照组n=40),利用ctDNA-WGBS技术检测从200μL血浆中获得的微量ctDNA(输入量约1ng)进行测序分析。研究结果发现,在多中心患者队列中,在早期和晚期BC阶段中对于15个ctDNA甲基化标志物的诊断特征表现出高准确性。同时可以在不同的癌症类型(包括肝细胞癌和肺癌)中可以很好区分雌激素受体状态的ctDNA甲基化特征。
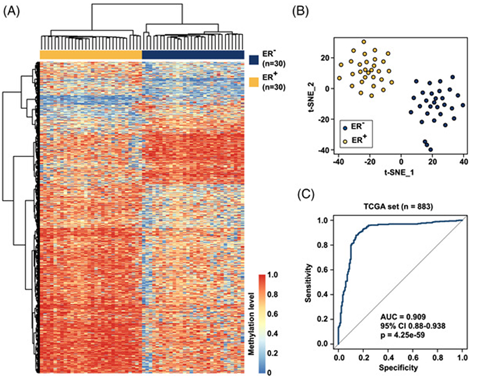
图:ctDNA甲基化可以作为乳腺癌亚型分类和预测ER状态的潜在生物标志物[3]
(2)MeDIP-seq结合机器学习方法,为多癌种生物标志物检测提供新思路。
此研究通过结合cfMeDIP-seq技术和机器学习,分析了215例样本的cfDNA甲基化组,包括患有肝癌或脑癌的个体样本(实验组)以及无癌症个体样本(对照组)。利用DMRs、DhMRs或DMRs+DhMRs训练机器学习模型,结果发现DMRs+DhMRs联合模型在区分对照组、肝癌和脑癌方面表现出更优性能,研究支持同时利用DMRs和DhMRs进行多癌种检测的潜力。

图:用于分析血浆cfDNA甲基化的单链cfDNA甲基化DNA免疫沉淀测序方法[4]
(3)ACE-seq无需亚硫酸盐转化精准区分甲基化和羟甲基化标记
此研究建立一种利用DNA脱氨酶(APOBEC3A)实现的非破坏性的、低起始DNA量的鉴定5hmC技术(ACE-seq)。与传统亚硫酸盐测序(BS-Seq)不同,ACE-seq不依赖于亚硫酸盐处理,而是通过酶促脱氨反应来区分未修饰的胞嘧啶(C)和5-甲基胞嘧啶(5mC),同时保护5hmC不被转化为尿嘧啶。这种方法允许对cfDNA中的5hmC进行精确的定位和定量分析。
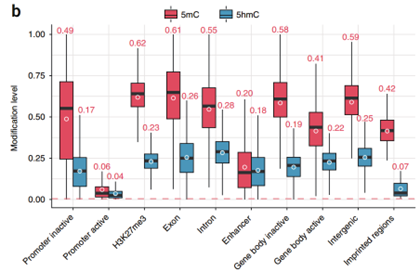
图:几类基因组元件的 5hmC(蓝色)和 5mC(红色)的修饰水平[2]
(4)cfhMeDIP-seq对5-羟甲基胞嘧啶(5hmC)特征进行检测以预测早期胰腺癌
此研究利用cfDNA hMeDIP-seq对胰腺导管腺癌(PDAC)队列(n=64)与非癌症队列(n=243)的5hmC变化进行比较分析。在数千个基因中鉴定差异性羟甲基化,结果表明胰腺发育或功能相关基因和癌症发病机制相关基因差异最为显著。在PDAC队列的cfDNA羟甲基化组中,与KRAS激活和TP53失活在PDAC肿瘤中常见基因调控失调相关的基因表现出差异性富集,表明5hmC变化在早期疾病阶段能实现PDAC分类。
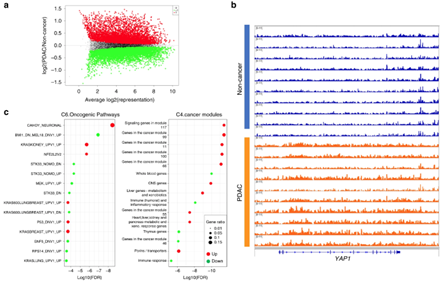
图:与非癌症患者cfDNA相比,PDAC cfDNA中的5hmC占位差异分析[5]
(5)cfChIP-seq利用cfDNA片段分析组蛋白修饰,揭示H3K27ac水平与cfDNA片段大小有关
此研究利用cfChIP-seq检测技术从18个健康对照样本中获得H3K27ac信号,并结合配对样本分析循环核小体的cfDNA片段大小模式和H3K27ac信号之间的相关性。随后构建线性回归模型,可根据cfDNA片段大小模式推断血浆中H3K27ac水平(H3K27ac相关信号)。分析发现H3K27ac相关信号与cfChIP-seq测定的H3K27ac相关信号一致。

图:cfChIP-seq揭示H3K27ac水平与cfDNA片段大小之间的关系[7]
(6) cfDNA低覆盖度WGS揭示肿瘤患者的全基因组突变特征
此研究对215名患者和227名健康人群的血浆进行全基因组测序(WGS),鉴定出病理性和生理性突变特征。
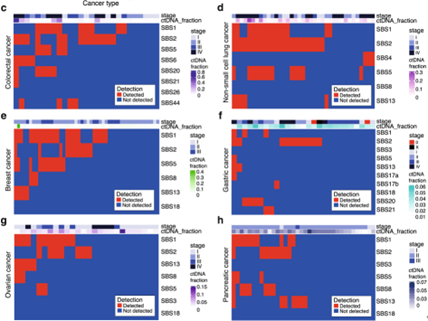
图:多种癌症类型的血浆基因突变特征检测[6]
参考文献:
① Shen SY, Burgener JM, Bratman SV, De Carvalho DD. Preparation of cfMeDIP-seq libraries for methylome profiling of plasma cell-free DNA. Nat Protoc. 2019 Oct;14(10):2749-2780. pii: 10.1038/s41596-019-0202-2. doi: 10.1038/s41596-019-0202-2. PubMed PMID: 31471598.
② Schutsky EK, et al.Nondestructive, base-resolution sequencing of 5-hydroxymethylcytosine using a DNA deaminase. Nat Biotechnol. 2018 Oct 8. pii: nbt.4204. doi: 10.1038/nbt.4204. PubMed PMID: 30295673.
③ Gao Y, et al. Whole-genome bisulfite sequencing analysis of circulating tumour DNA for the detection and molecular classification of cancer. Clin Transl Med. 2022 Aug;12(8):e1014. doi: 10.1002/ctm2.1014. PubMed PMID: 35998020.
④ Hua X, et al. Tumor detection by analysis of both symmetric- and hemi-methylation of plasma cell-free DNA. Nat Commun. 2024 Jul 20;15(1):6113. pii: 10.1038/s41467-024-50471-1. doi: 10.1038/s41467-024-50471-1. PubMed PMID: 39030196.
⑤ Guler GD,et al. Detection of early stage pancreatic cancer using 5-hydroxymethylcytosine signatures in circulating cell free DNA. Nat Commun. 2020 Oct 19;11(1):5270. pii: 10.1038/s41467-020-18965-w. doi: 10.1038/s41467-020-18965-w. PubMed PMID: 33077732.
⑥ Wan JCM, et al. Genome-wide mutational signatures in low-coverage whole genome sequencing of cell-free DNA. Nat Commun. 2022 Aug 23;13(1):4953. pii: 10.1038/s41467-022-32598-1. doi: 10.1038/s41467-022-32598-1. PubMed PMID: 35999207.
⑦ Bai J, et al. Histone modifications of circulating nucleosomes are associated with changes in cell-free DNA fragmentation patterns. Proc Natl Acad Sci U S A. 2024 Oct 15;121(42):e2404058121. doi: 10.1073/pnas.2404058121. PubMed PMID: 39382996.
