单细胞甲基化+转录组测序(scBS+RNA-seq)
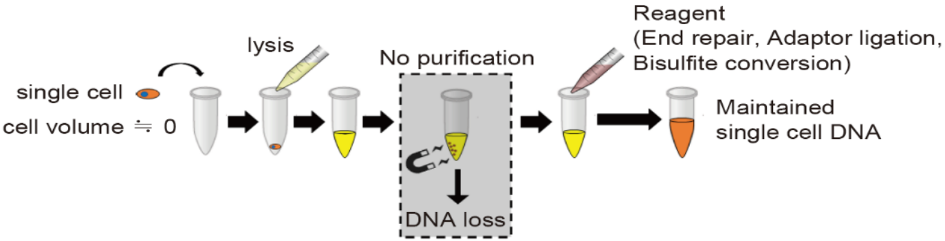
与常规RRBS不同,高通量单细胞甲基化测序sc-RBS由于从单个细胞输入的DNA量很少,常规方法不能保证目标DNA在最后的扩增过程中保持不变,因此sc-RBS在降损方面进行细化。易基因建立的高通量单细胞甲基化测序sc-RBS技术无需纯化,通过限制性内切酶消化、接头连接过程和在单管中转化重亚硫酸盐,是一种专注于将纯化过程中发生的损失降至最低的方法。
应用方向
5、性价比高:单个细胞检测费用低至千余元。
技术路线
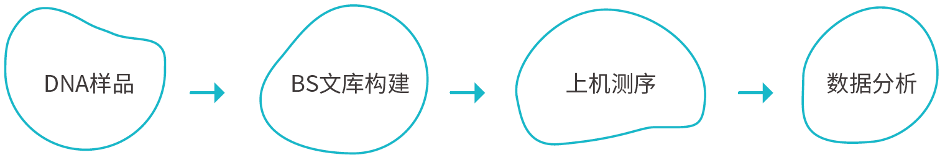
实验策略
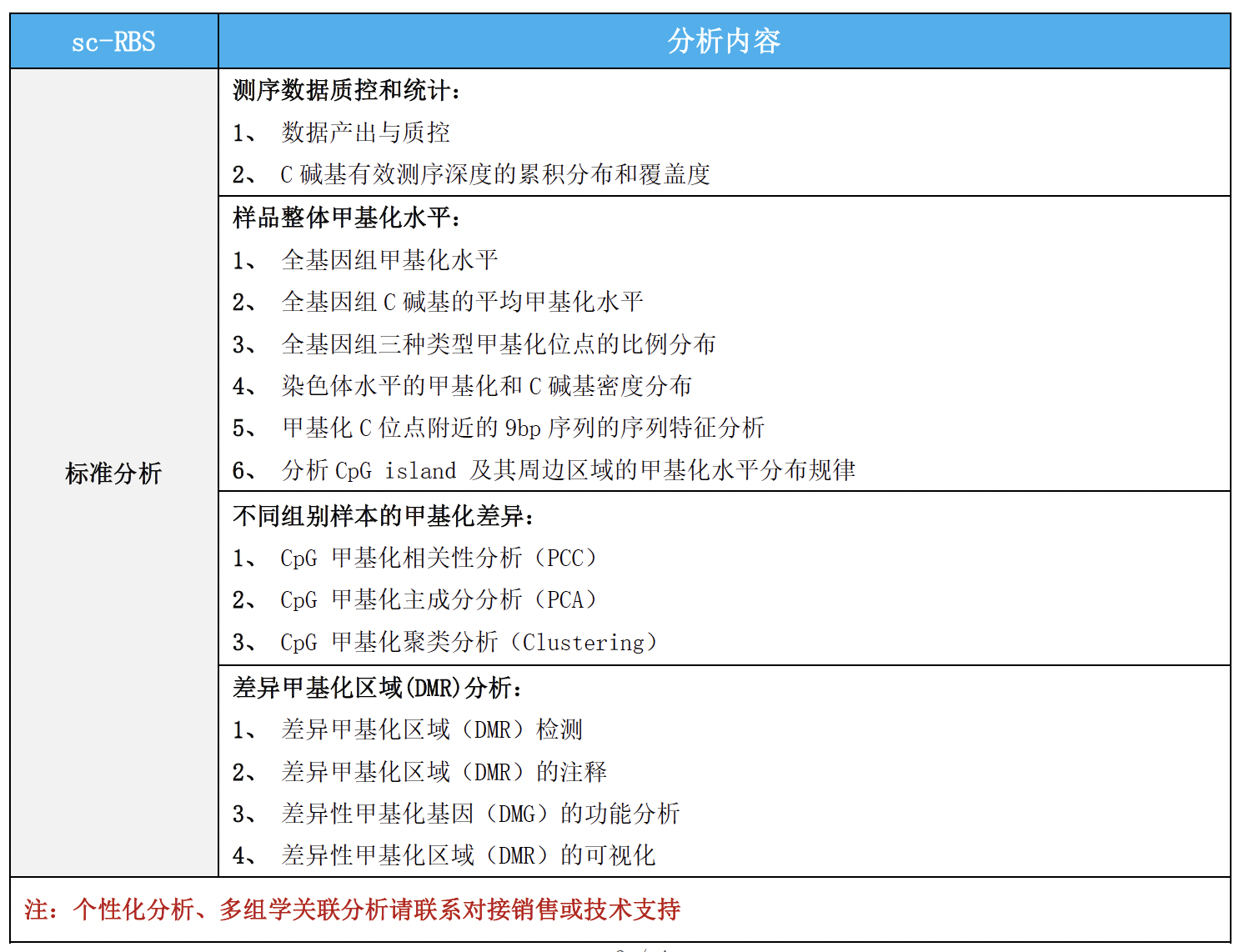
分析内容
送样要求

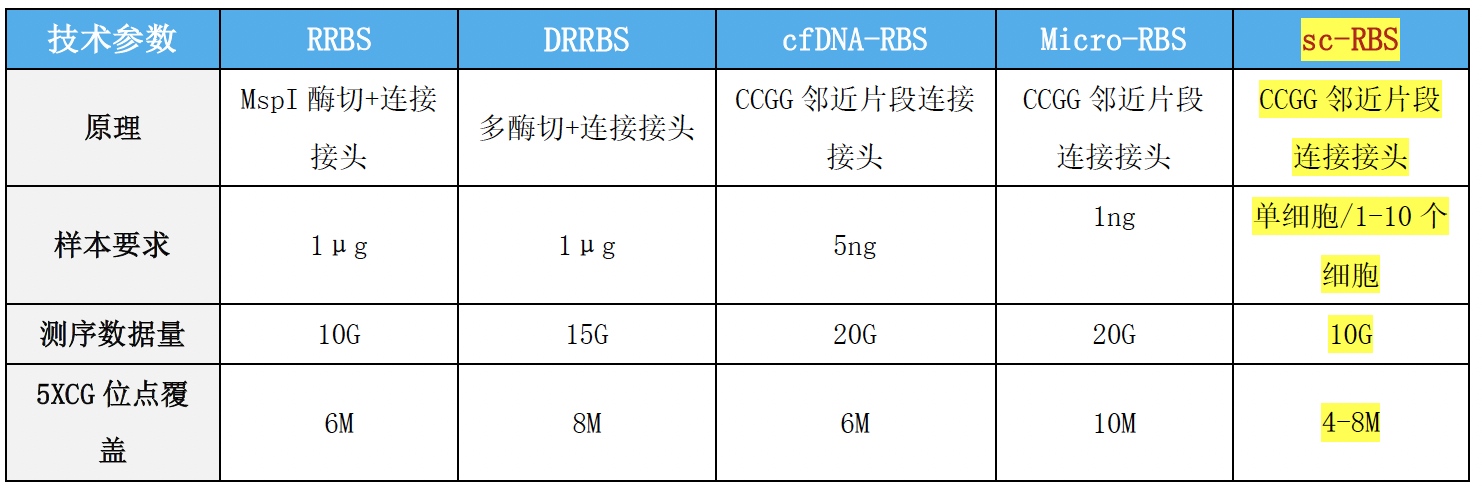
技术指标
经典案例
sc-RBS+RNA-seq测序揭示黄曲霉毒素B1诱导S期阻滞L02细胞肝毒性新机制
Single-cell sequencing reveals novel mechanisms of Aflatoxin B1-induced hepatotoxicity in S phase-arrested L02 cells
1.背景:
大量研究证据表明黄曲霉毒素B1(Aflatoxin B1 ,AFB1)诱导的毒性包括抑制细胞增殖、氧化应激、DNA损伤、细胞周期阻滞和细胞凋亡,特别是针对肝脏细胞具有强烈的毒性。但AFB1毒性的表观遗传机制目前尚不清楚,需要进一步探讨。
2.方法:
本研究通过单细胞测序技术(scRNA-seq + sc-RRBS)研究AFB1诱导S期阻滞L02细胞肝毒性机制。
3.结论:
单细胞测序技术结果发现AFB1可诱导细胞凋亡和细胞周期S期阻滞,降低线粒体膜电位(ΔΨm),增加活性氧(ROS)生成。通过促性腺激素释放激素受体通路,Wnt信号传导通路,TGF-β信号通路等调节DNA甲基化水平。研究结果揭示了AFB1对S期阻滞L02细胞诱导的肝毒性机制,其中DNA甲基化通过调控促性腺激素释放激素受体通路、Wnt信号传导通路和TGF-β信号通路发挥作用,为进一步研究精确毒理学提供了新的探索策略,包括靶细胞选择、多组非定向测序和通路分析。
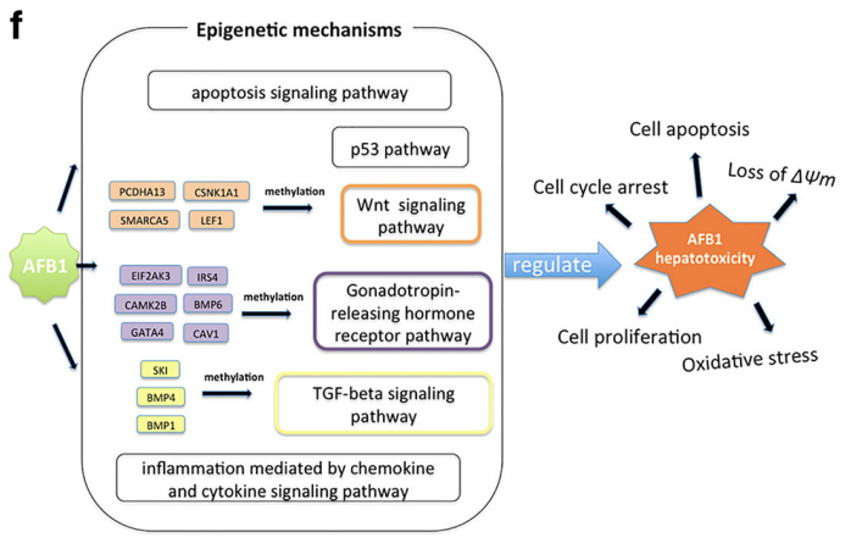
图:基于单细胞测序结果,研究人员提出AFB1诱导的S期阻滞L02细胞肝毒性调节机制的假设
