RNA免疫共沉淀测序 (RIP-seq)
RIP-seq是将RNA免疫共沉淀(RNA Immunoprecipitation,RIP)与二代测序技术(NGS)相结合以研究细胞内RNA与蛋白互作的技术,RIP利用目标蛋白抗体把相应的RNA-蛋白复合物(RNA Binding Protein,RBP)沉淀下来,然后经过富集和纯化就可以对结合在复合物上的RNA进行测序分析。
RIP-seq对富集得到的RNA片段通过高通量测序,在全转录组范围内对细胞内RNA与蛋白结合情况进行分析,揭示RNA分子与RBP互作(包括非编码RNA与蛋白互作)。RIP-seq是了解转录后调控网络动态过程的有力工具,能更有效地发现miRNA的调控靶点。
应用领域:
1)全转录组范围内揭示RNA分子与RBP互作
2)RNA与靶蛋白相互作用的验证
3)RBP与mRNA的互作分析
技术优势:
1)全转录组覆盖:可在全转录组范围对蛋白结合位点进行筛选与鉴定
2)高灵敏度:每个样本可获得数百万条的序列标签,检测转录本上更多的蛋白结合位点
3)高精确率:可获得高水平的信噪比数据,准确区分真实事件与噪音
4)多种商品化抗体,高效支持实验开展
实验策略:

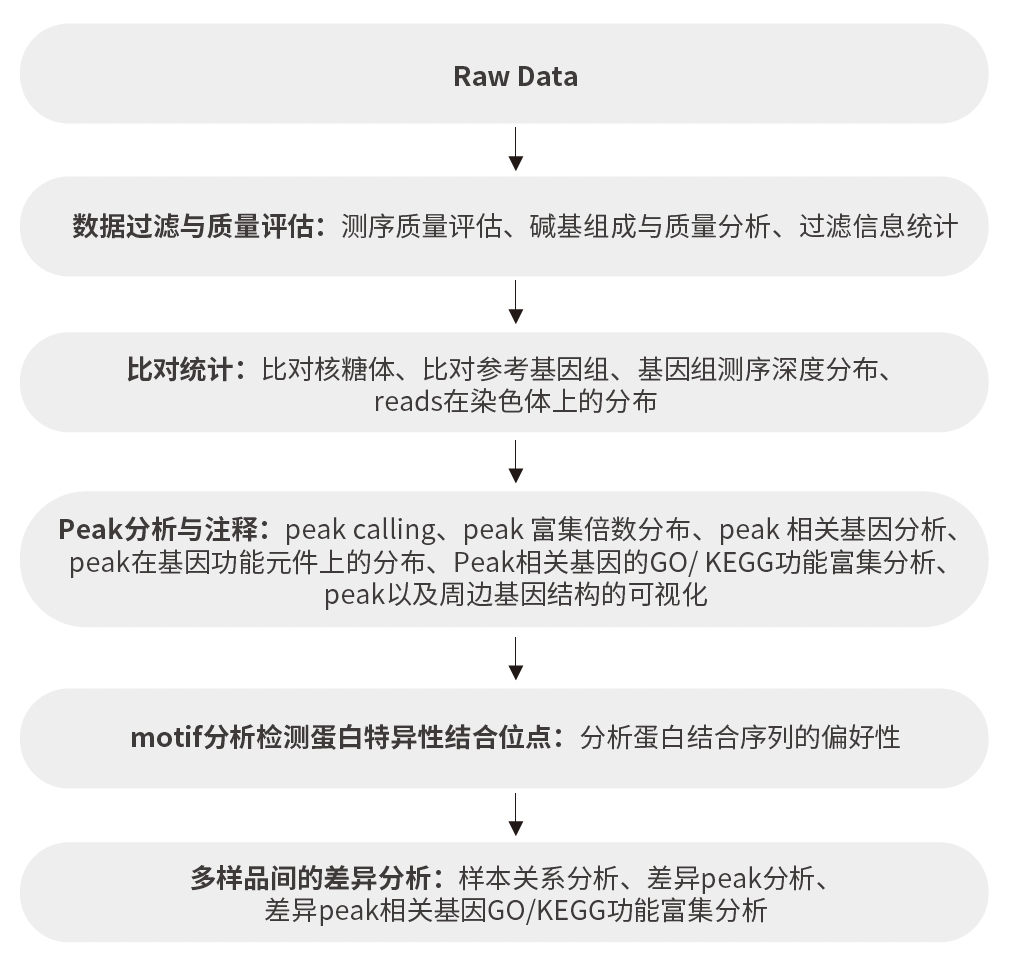
分析内容:
送样要求:

案例解析:
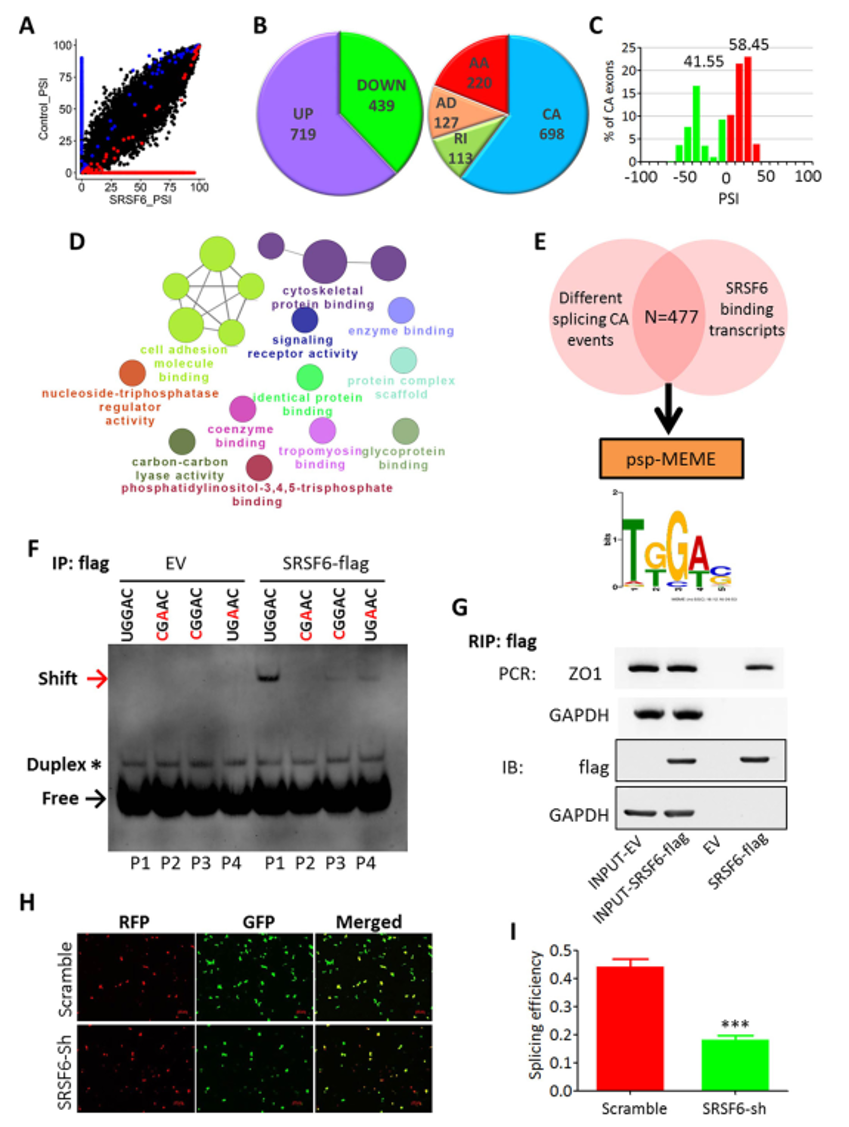
对311个CRC样本、癌症基因组图谱和基因表达综合数据库(GEO)数据库中SRSF6的表达进行全面分析。通过RIP-seq鉴定SRSF6调控的可变剪接(AS)及其结合位点,并通过凝胶迁移和微基因报告实验进行验证。
结果: