EWAS(Epigenome-Wide Association Study)全基因组关联研究是一种探索表观遗传变异与特定疾病或表型之间关系的研究方法,通常涉及DNA甲基化水平的全基因组分析,以鉴定与疾病相关的甲基化位点。EWAS是筛选与疾病相关的甲基化位点和区域的强大工具。
EWAS研究思路
主线研究:通过EWAS分析寻找与表型相关的甲基化位点。
组学关联:结合甲基化数据与SNP数据,进行meQTL分析,Finemap精细定位,以及与转录组数据结合,分析差异甲基化位点与基因表达之间的相关性。
因果探索:进一步判断差异甲基化位点是否对表型起因果决定作用,可以进行因果推断检验(CIT)分析。
EWAS研究方案
1. 研究目的与假设
目的:探索特定疾病或表型与DNA甲基化水平之间的关联,以鉴定与疾病相关的甲基化位点。
假设:特定的DNA甲基化位点与特定疾病或表型存在显著相关性。
2. 样本收集与制备
样本类型:根据研究目的选择合适的生物样本,如全血、唾液、组织等。
样本量:参考JAMA Psychiatry的研究,至少需要数千例样本以确保统计效力。
样本处理:确保样本的采集、存储和处理符合伦理和科学标准。
3. DNA甲基化水平测定
技术平台:选择合适的高通量测序平台,如RRBS、甲基化芯片。
数据获取:对每个样本进行DNA甲基化水平检测,获取全基因组甲基化数据。
4. 数据预处理
质量控制:对原始数据进行质量控制,排除低质量的样本和探针。
标准化:对甲基化数据进行标准化处理,以消除技术批次效应。
5. 统计分析
差异甲基化分析:使用统计软件(如R语言中的minfi或DSS包)进行差异甲基化分析。
关联分析:对差异甲基化位点进行关联分析,确定与疾病或表型相关的甲基化位点。
6. 组学关联分析
meQTL分析:结合甲基化数据与SNP数据,进行meQTL分析,以识别甲基化QTL。
Finemap精细定位:对关联信号进行精细定位,以提高定位精度。
基因表达关联分析:结合转录组数据,分析差异甲基化位点与基因表达之间的相关性。
7. 因果探索
因果推断检验(CIT):对差异甲基化位点进行因果推断检验,以判断其对表型的因果作用。
8. 功能富集分析
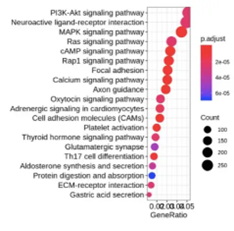
KEGG富集分析:对靶基因进行KEGG功能富集分析,以识别生物学途径和功能模块。
9. 结果验证
独立样本验证:使用独立样本集对发现的甲基化位点进行验证。
EWAS图示
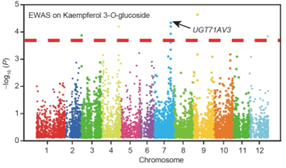
EWAS曼哈顿图
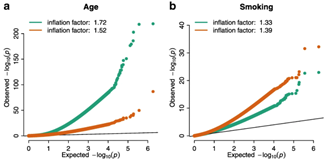
EWAS研究QQ图
将EWAS分析所得到的靶基因进行KEGG功能富集
EWAS典型案例
(1)JAMA psychiatry:DNA甲基化发现抑郁症潜在的分子标记物
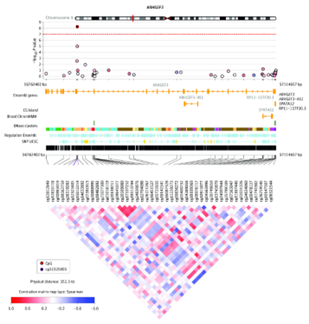
抑郁症发现阶段7948例,验证阶段3308例,共11256例受试者的全血样本进行全表观基因组关联分析(EWAS)。在抑郁症发现阶段,对7948例受试者的全血样本进行EWAS,鉴定出显著差异的甲基化位点cg04987734。在验证阶段进一步验证3个与抑郁症相关的甲基化位点,这些位点不仅有望成为抑郁症的生物标志物,还可能揭示病理机制及临床治疗的潜在作用。
图:显著相关甲基化位点cg12325605的EWAS分析结果模式图
(2)苏格兰万人队列表观基因组关联研究揭示DNA甲基化与多种疾病之间的关系
研究通过对单个族裔大样本队列--18413个苏格兰志愿者全血样本的752722个CpG甲基化位点进行分析,评估19种疾病与DNA甲基化的相关性。在分析DNA甲基化与14种不同疾病相关性后,进一步分析19种疾病的发病分析,通过年龄、性别、血细胞计数校正后揭示11种疾病里的14237个显著相关位点,其中分别有11305个和2657个位点与慢阻肺和2型糖尿病相关。进一步纳入生活方式与人群结构校正后,发现5种疾病的79个位点相关。交叉对比分析揭示2种疾病的64个相关,其中2型糖尿病(58个CpG)、慢阻肺(6个CpG)。
图:19种疾病的发病分析关联结果以及2种疾病的EWAS展示
参考文献:
1. Michels K B, Binder A M, Dedeurwaerder S, et al. Recommendations for the design and analysis of epigenome-wide association studies[J]. Nature methods, 2013, 10(10): 949-955.
2. Story Jovanova O, et al. DNA Methylation Signatures of Depressive Symptoms in Middle-aged and Elderly Persons: Meta-analysis of Multiethnic Epigenome-wide Studies. JAMA Psychiatry. 2018 Sep 1;75(9):949-959. pii: 2687369.
3. Hillary RF, et al. Blood-based epigenome-wide analyses of 19 common disease states: A longitudinal, population-based linked cohort study of 18,413 Scottish individuals. PLoS Med. 2023 Jul 6;20(7):e1004247. doi: 10.1371/journal.pmed.1004247. PMID: 37410739; PMCID: PMC10325072.